DNAは、炭素、水素、酸素、窒素、及びリン原子から構成される典型的な有機化合物です。しかし、DNAは他の有機化合物とは大きく違い、酵素によって増幅が可能です。この酵素によるDNAの増幅はポリメラーゼ連鎖反応(PCR)と呼ばれ、現在は遺伝子検出に幅広く使用されています。一方、我々はDNAのバルジ構造と呼ばれる相補的な塩基が無く、フリーの塩基が残っている構造に特異的に結合し、特徴的な蛍光を発する蛍光小分子(N,N’-bis(3-aminopropyl)-2,7-diamino-1,8-naphthyridine (DANP))を発見しました。このDANPは1)DNAのシトシンバルジ構造に特異的に結合する、以外にも2)DNAの二重鎖には結合しない、3)シトシンバルジに結合すると分子本来の吸収及び蛍光スペクトルより長波長側に新たなスペクトルが生じるという特徴を持っています。この特徴を使って、安価で迅速、高感度の遺伝子蛍光検出法の開発を行いました。
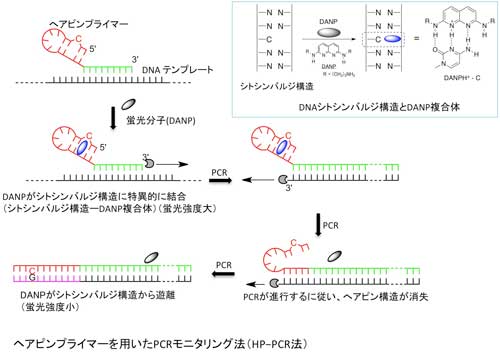
現在汎用されているPCR法はDNA2重鎖を検出するSYBR® Green法とTaqMan®法があります。前者はDNAを増幅して得られた2重鎖を検出する方法であり、簡便ではありますが、非特異的増幅による二本鎖も検出してしまいます。そのため特異的増幅産物の検出には、より高価なTaqManプローブを使う後者の方法が必要になります。一方、ヘアピンプライマー法ではヘアピン構造で標識されたプライマーの消費だけをモニターするために、非特異的な増幅に対しても簡単に対応できます。このヘアピンプライマーPCR法を使って、シトクロム P450のサブタイプ(2C9*3)の遺伝子検出を、野生型と変異型にマッチするプライマーを用いて行った結果、野生型と変異型では蛍光変化に明らかな差が生じ、一塩基の違いを検出することが出来ました。
現在はこの方法をウイルス検出に応用し、ウイルスの変異にも迅速に対応できる検出法の開発を目指しています。
(1) Chen, H.; Takei, F.; Koay, E. S.-C.; Nakatani, K.; Chu, J. J. H. J. Mol. Diagn. 2013, 15, 227-233. (2) Takei, F.; Igarashi, M.; Oka, Y.; Koga, Y.; Nakatani, K. ChemBioChem 2012, 13, 1409-1412. (3) Takei, F.; Igarashi, M.; Hagihara, M.; Oka, Y.; Soya, Y.; Nakatani, K. Angew. Chem. Int. Ed. 2009, 48, 7822-7824. (4) Takei, F.; Suda, H.; Hagihara, M.; Zhang, J.; Kobori, A.; Nakatani, K. Chem. Eur. J. 2007, 13, 4452-4457.
|